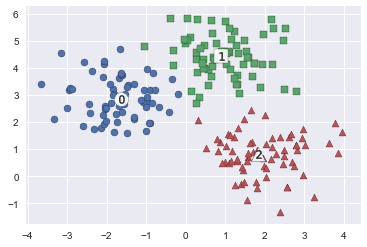
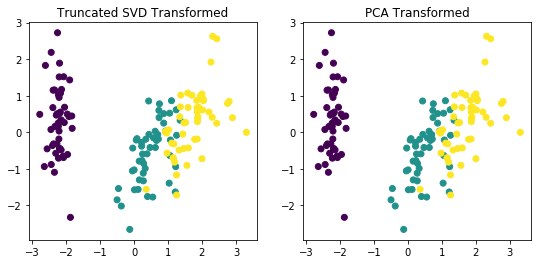GMM 을 이용한 붓꽃 데이터 셋 클러스터링
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
%matplotlib inline
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
# 보다 편리한 데이타 Handling을 위해 DataFrame으로 변환
irisDF = pd.DataFrame(data=iris.data, columns=feature_names)
irisDF['target'] = iris.target
GaussianMixture를 이용하여 붓꽃 데이터 군집화
from sklearn.mixture import GaussianMixture
gmm = GaussianMixture(n_components=3, random_state=0).fit(iris.data)
gmm_cluster_labels = gmm.predict(iris.data)
# 클러스터링 결과를 irisDF 의 'gmm_cluster' 컬럼명으로 저장
irisDF['gmm_cluster'] = gmm_cluster_labels
# target 값에 따라서 gmm_cluster 값이 어떻게 매핑되었는지 확인.
iris_result = irisDF.groupby(['target'])['gmm_cluster'].value_counts()
print(iris_result)
target gmm_cluster
0 0 50
1 2 45
1 5
2 1 50
Name: gmm_cluster, dtype: int64
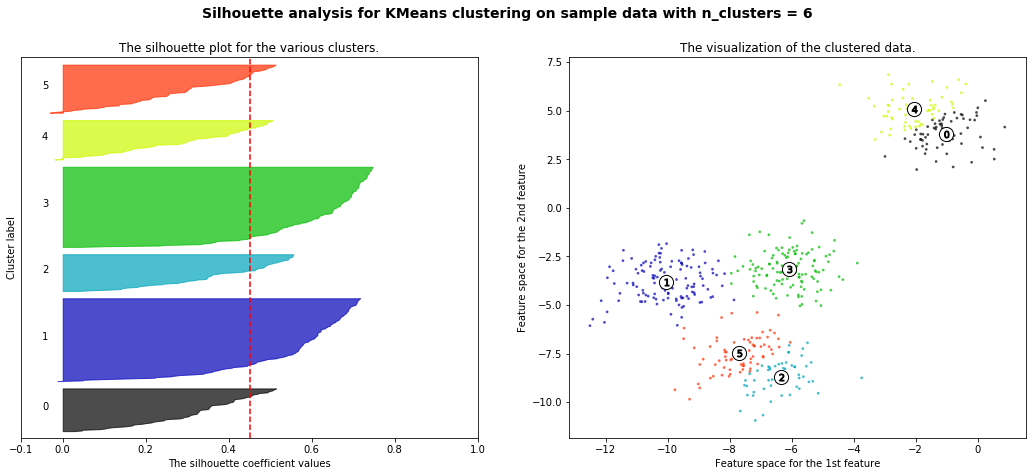
붓꽃 데이터 K-Means 군집화 결과
iris.target_names
# array(['setosa', 'versicolor', 'virginica'], dtype='<U10')
kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=300,random_state=0).fit(iris.data)
kmeans_cluster_labels = kmeans.predict(iris.data)
irisDF['kmeans_cluster'] = kmeans_cluster_labels
iris_result = irisDF.groupby(['target'])['kmeans_cluster'].value_counts()
print(iris_result)
target kmeans_cluster
0 1 50
1 0 48
2 2
2 2 36
0 14
Name: kmeans_cluster, dtype: int64
클러스터링 결과를 시각화하는 함수 생성
### 클러스터 결과를 담은 DataFrame과 사이킷런의 Cluster 객체등을 인자로 받아 클러스터링 결과를 시각화하는 함수
def visualize_cluster_plot(clusterobj, dataframe, label_name, iscenter=True): # 군집중심좌표가 GMM에 없어서 iscenter 해주기
if iscenter :
centers = clusterobj.cluster_centers_
unique_labels = np.unique(dataframe[label_name].values)
markers=['o', 's', '^', 'x', '*']
isNoise=False
for label in unique_labels:
label_cluster = dataframe[dataframe[label_name]==label]
if label == -1: # -1은 db스캔에서 노이즈
cluster_legend = 'Noise'
isNoise=True
else :
cluster_legend = 'Cluster '+str(label)
plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], s=70,\
edgecolor='k', marker=markers[label], label=cluster_legend)
if iscenter: # 시각화 코드
center_x_y = centers[label]
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=250, color='white',
alpha=0.9, edgecolor='k', marker=markers[label])
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k',\
edgecolor='k', marker='$%d$' % label)
if isNoise:
legend_loc='upper center'
else: legend_loc='upper right'
plt.legend(loc=legend_loc)
plt.show()
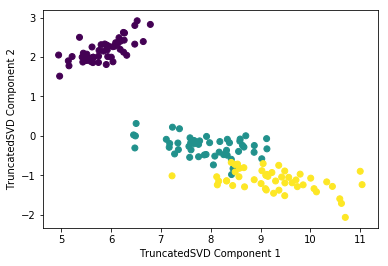
GMM군집화와 K-Means군집화를 비교하기 위해 타원형으로 늘어선 임의의 데이터 세트를 생성
from sklearn.datasets import make_blobs
# make_blobs() 로 300개의 데이터 셋, 3개의 cluster 셋, cluster_std=0.5 을 만듬.
X, y = make_blobs(n_samples=300, n_features=2, centers=3, cluster_std=0.5, random_state=0)
# 길게 늘어난 타원형의 데이터 셋을 생성하기 위해 변환함.
transformation = [[0.60834549, -0.63667341], [-0.40887718, 0.85253229]]
X_aniso = np.dot(X, transformation)
# feature 데이터 셋과 make_blobs( ) 의 y 결과 값을 DataFrame으로 저장
clusterDF = pd.DataFrame(data=X_aniso, columns=['ftr1', 'ftr2'])
clusterDF['target'] = y
# 생성된 데이터 셋을 target 별로 다른 marker 로 표시하여 시각화 함.
visualize_cluster_plot(None, clusterDF, 'target', iscenter=False)
K-Means 군집화 수행
# 3개의 Cluster 기반 Kmeans 를 X_aniso 데이터 셋에 적용
kmeans = KMeans(3, random_state=0)
kmeans_label = kmeans.fit_predict(X_aniso)
clusterDF['kmeans_label'] = kmeans_label
visualize_cluster_plot(kmeans, clusterDF, 'kmeans_label',iscenter=True)
GMM 군집화 수행
# 3개의 n_components기반 GMM을 X_aniso 데이터 셋에 적용
gmm = GaussianMixture(n_components=3, random_state=0)
gmm_label = gmm.fit(X_aniso).predict(X_aniso)
clusterDF['gmm_label'] = gmm_label
# GaussianMixture는 cluster_centers_ 속성이 없으므로 iscenter를 False로 설정.
visualize_cluster_plot(gmm, clusterDF, 'gmm_label',iscenter=False)
GMM과 K-Means 군집화 결과 비교
print('### KMeans Clustering ###')
print(clusterDF.groupby('target')['kmeans_label'].value_counts())
print('\n### Gaussian Mixture Clustering ###')
print(clusterDF.groupby('target')['gmm_label'].value_counts())
### KMeans Clustering ###
target kmeans_label
0 2 73
0 27
1 1 100
2 0 86
2 14
Name: kmeans_label, dtype: int64
### Gaussian Mixture Clustering ###
target gmm_label
0 2 100
1 1 100
2 0 100
Name: gmm_label, dtype: int64
'Data_Science > ML_Perfect_Guide' 카테고리의 다른 글
| 7-9. DBSCAN 2 (0) | 2021.12.30 |
|---|---|
| 7-8. DBSCAN (0) | 2021.12.30 |
| 7-6. Mean_Shift (0) | 2021.12.30 |
| 7-4. KDE(Kernel Density Estimation) (0) | 2021.12.30 |
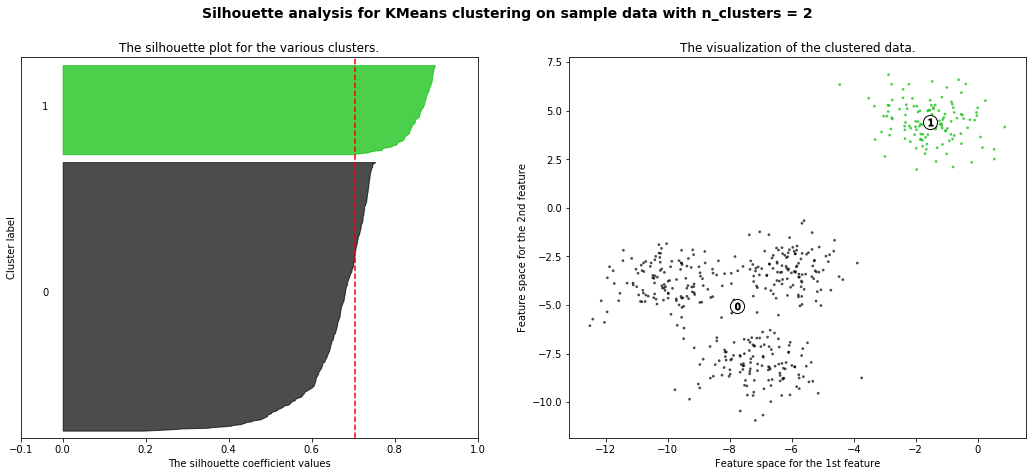
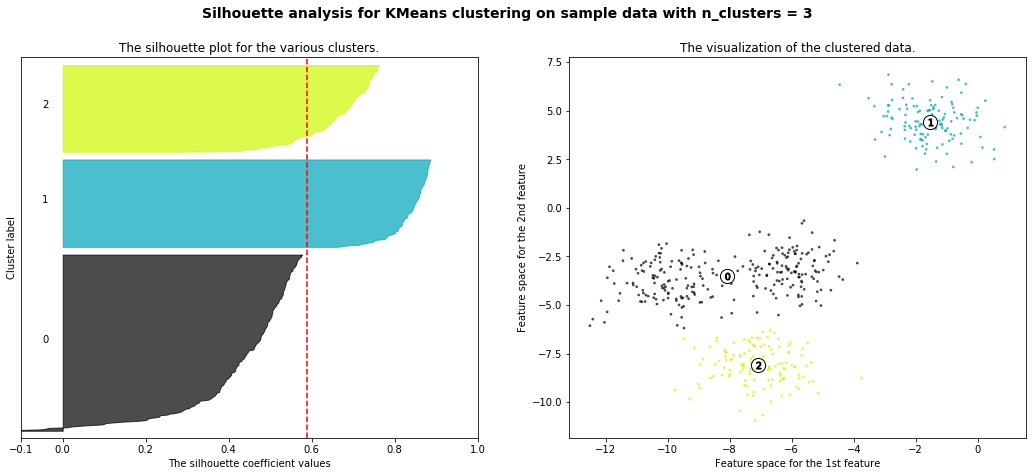
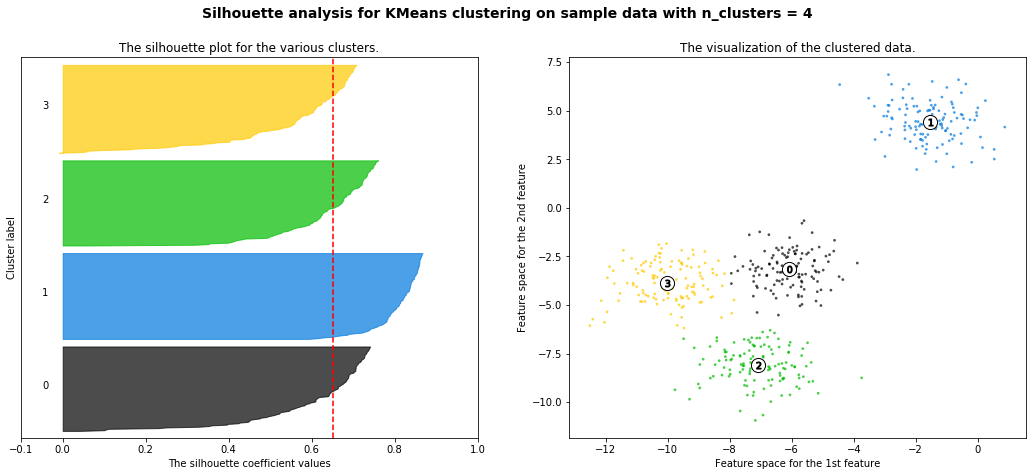
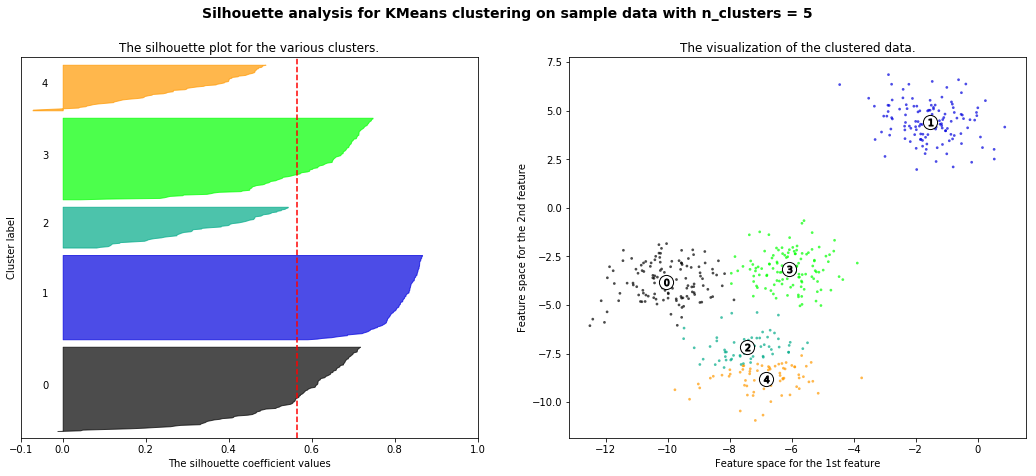
| 7-3. Cluster_evaluation || 실루엣 계수 (0) | 2021.12.30 |